
MOE新版本发布啦!更强的数据显示,更快的运算速度,更全面的模拟功能!新版本功能更新如下:
硬件更新:
√ 支持Apple M1芯片:安装MOE前需要用户下载XQuartz 2.8.0(Xquartz 2.8.1存在文本渲染性能漏洞,暂不支持)

√ 支持GPU加速:目前MOE2022.02的GPU加速需要支持cuda10.0版本以上的GPU及NVIDIA显卡驱动。
功能更新:
√ mdb显示及数据处理:
增强mdb中的数据显示:碳水化合物可以以SNFG符号显示、蛋白及核酸可以以FASTA序列显示,另增加分子手性显示、3D视图匹配及图片显示功能
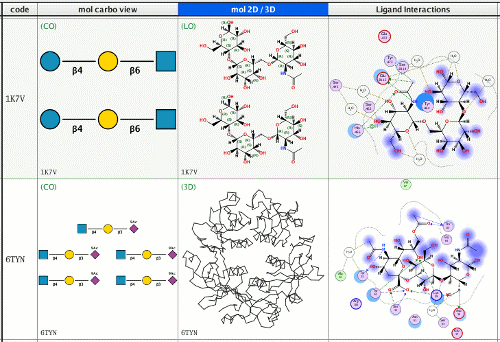
增强mdb中的数据搜索:支持组合搜索、多字段搜索、通配符搜索等
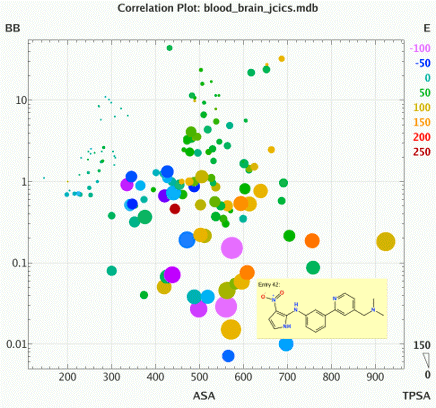
增强mdb中的数据绘图:支持x/y轴的缩放、数据点结构显示等
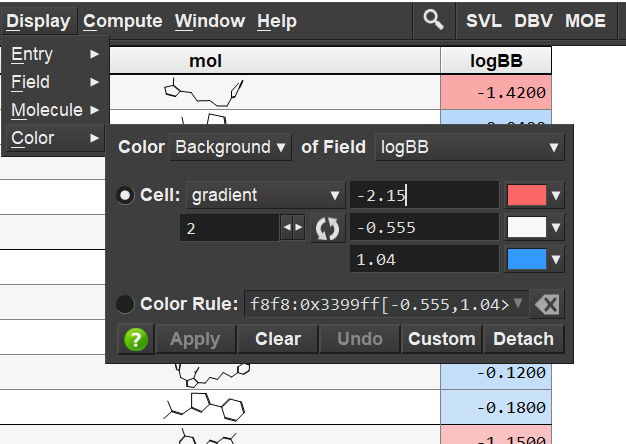
增强mdb中的数据着色:支持梯度着色和对数梯度着色、自定义规则着色等
√ 支持GPU加速的功能:
Sidechain packing:利用一元二次优化(UQO)技术的大规模并行化部分来高效地搜索旋转异构体的最佳组合,并在GPU上重新实现,包含侧链打包的模块有:Protein Design, Homology Model, Antibody Modeler, Loop Modeler, Protein Builder, Protein-Protein Dock。侧链放置速度增加90倍,蛋白质建模,整体速度提高40%。
蛋白-蛋白对接:在粗粒化模型最小化步骤中,GPU重新实现了对粗粒模型相互作用能的计算,其速度提高了6 - 10倍。
√ 抗体模建功能的优化:
抗体模建通过连接两个Fv (VL-linker-VH, VH-linker-VL)构建scFv,其中连接片段需要是查询序列的一部分。得到的scFv结构可以嫁接到完整或片段抗体模板上。
√ moeweb添加了反应式绘制功能:
在反应模式下,可以上传、显示和编辑反应。支持导入MOL、RXN、Reaction SMILES三种格式。
反应物和产物原子之间的编号映射可以自动生成或手动指定。
为了更容易地识别反应中改变的键,在反应物中断裂的键和在产物中形成的键用两条平行线标出,颜色与试剂匹配。
增强了原子查询注释,添加了新的原子通配符和查询属性
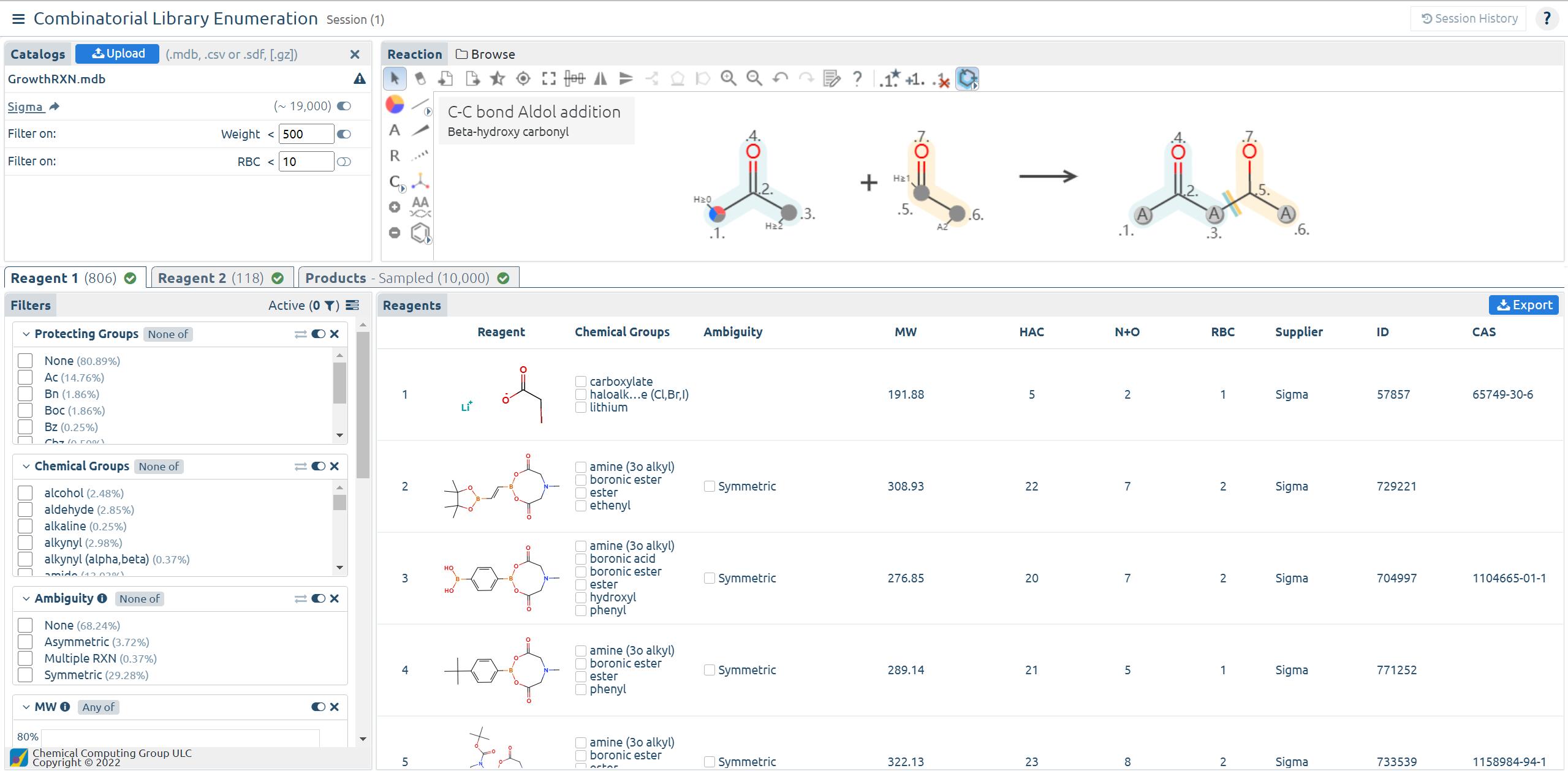
√ 组合库枚举:组合库枚举(CLE) web应用程序是一个基于反应的库枚举器。它使用了一种新的基于概率的虚拟化合物枚举算法,允许交互式地浏览和优化整个枚举库,而不必实际完全枚举库。
支持上传各种格式的反应物目录。自定义供应商反应物库用于枚举,自动搜索与反应匹配的反应物。
根据分子性质和常见的化学保护基团,通过自定义筛选反应物和产物,调整库的内容。
自动分析反应物和产物性能。通过检查属性过滤器中的属性直方图,可视化整个库中属性的分布。通过对不断增长的枚举产物的动态累积统计来估计整个库的属性分布。
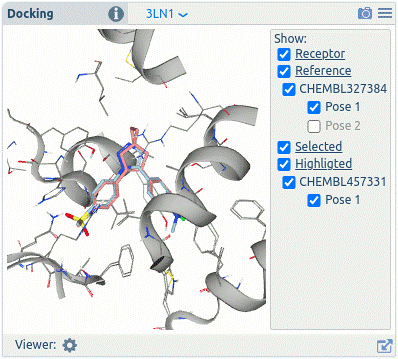
√ MOEsaic新增基于网页端的分子对接功能:
新增的窗口,具有3D显示区,用于可视化对接构象,并有一个侧边栏,可以选择显示或隐藏什么,并列出排队或正在进行的计算。
支持药效团对接,允许并行计算
在Sketch模式下,当分子进行实时修饰时,相应的对接计算是动态更新的。在MMP模式下,参考和显示的结构都会自动对接。
另外,数据库、描述符、SVL函数等均有更新,还有诸多实用功能,更详细的新功能介绍请访问:https://www.chemcomp.com/release_notes/moe202202/rnotes.htm


