我们很高兴通知您MOE新版本2019.01已经发布。MOE新版本中的功能和应用有比较大的更新,主要包括以下内容:
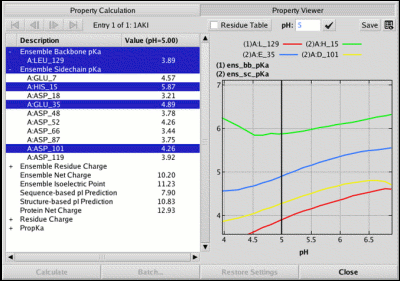
随pH依赖的蛋白质属性的计算和分析
(1)支持动态构象采样以计算多个构象下的蛋白属性;
(2)可以在单个pH环境或者一段范围的pH环境下计算描述符;
(3)计算条件,包括温度、盐浓度及类型均可自定义。
MOEsaic功能更新:项目分享和项目定制化
(1)目前,可以支持两种类型的定制化模型:Profile(类似于内置的Oral Drugs模型)和Scatter(类似于内置的Sweet Spot和Golden Triangle模型);
(2)实时项目可以通过一个API来自动进行交互式的更新;
(3)可支持分享项目副本或者只读的交互式快照;
(4)MMP分析更新,可支持MMP Profile Plot,直接进行结构间比较。
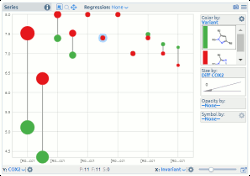
Custom MOEsaic model MMP Profile plot: COX2 activity vs. invariant
支持从NMR NOE数据分析构象分布
(1)增加色谱分析功能,提供分析液相中首选构象;
(2)提供3种可操作的模式:Conformers, Subset and Labels
(3)支持3种类型的实验数据:NMR shift data, NOE distance data和J-coupling data。

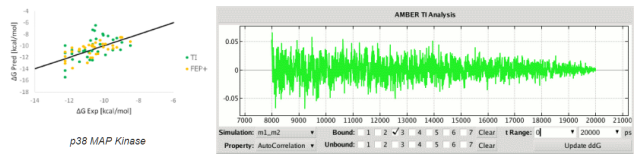
可使用AMBER Thermodynamic Integration进行相对结合自由能计算
(1)AMBER TI 可以在Amber 18版本的GPU中运算;MOE提供简化版的GPU AMBER TI计算的设置界面;
(2)MOE可以直接产生在集群或云服务器中进行AMBER TI计算的脚本,并且可以任务管理系统提交计算任务;
(3)开发AMBER TI分析面板,可直接在TI计算输出的database中进行分析。