
"MOE 2014.09版本新功能介绍"
内容简介:
MOE总部加拿大CCG公司于2014年12月9日正式发布了MOE新版本2014.09。MOE 2014.09引入了“基于结构的药物设计”的项目管理功能,能够管理SBDD 项目中的结构、序列、表面图等众多和SBDD 相关的数据。对接方面现在允许通过基于原有配体为模板的对接方法。新版本在构象生成后可以通过QM 进行精细的能量优化。在大分子设计方面的更新包括:抗体数据库生成、非天然氨基酸设计、蛋白结构家族数据库、模拟突变结果分析等等。
新特性:
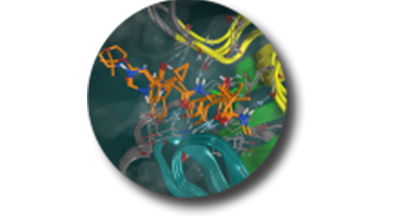

通过自动化的流程创建SBDD项目数据库
自动对SBDD项目中的结构、序列、表面图以及描述符等进行比对
可以在SBDD项目数据中收集家族结构数据,并在该家族结构数据中进行结构模建和loop构象采样


计算特定氨基酸的突变频率和概率
通过突变概率打分来降低突变空间
通过综合计算稳定性和亲和力来确定有利突变
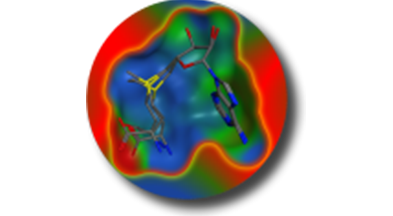

通过MM快速生成构象并通过QM的方法进行优化
重新设计的功能更强的Gaussian接口
通过MOE/web SOAP的QM功能快速精确的计算构象和能量


选择原有配体的部分结构作为模板进行对接
基于配体相似度或进行最大子结构匹配的对接
可以在一个数据库中进行结构叠合
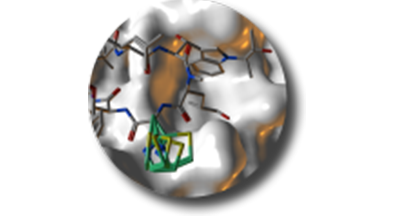

含有非天然氨基酸残基的蛋白的构建及模拟
构建非天然氨基酸残基并进行构象搜索
模拟并虚拟优化非天然氨基酸残基进行ADC设计
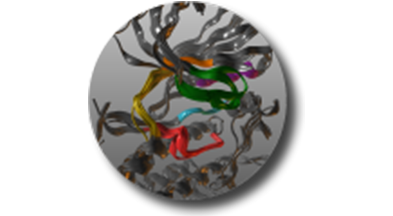

更多独有的蛋白结构家族数据库 (GPCR, Kinase, etc.) 并且里面包含了私有的结构数据
在特定的蛋白家族数据库中进行结构、序列和配体相似度的搜索
在大量的复合物数据库中进行蛋白-配体相互作用指纹图谱的计算


