
 2020-04-01
2020-04-01
 康昱盛
康昱盛
上海康昱盛在多组学分析领域拥有多年积累经验,长期致力于提供前沿的基因组学,蛋白组学,代谢组学和脂质组学优质软件解决方案。在这个特殊时期,康昱盛希望能够为国内新冠相关研究提供力所能及的帮助,联合凯杰公司为中国正在进行SARS-CoV-2病毒及COVID-19疾病研究的科研工作者提供:
CLC Genomics Workbench & Ingenuity Pathway Analysis(IPA)限时免费!
▲ 国际主流的二代测序分析综合分析平台CLC Genomics Workbench提供基因组,转录组,表观基因组和宏基因组的综合数据分析。
CLC可以提供快速准确地组装新冠病毒全基因组、基因功能注释及遗传进化的一键式分析,为深入了解病毒的和零号病人发现的基因遗传特性,得到病毒的进化根源以及和其它病毒的关系。特别是在SARS-CoV-2广泛传播过程中,发生突变位点的检测,构建病毒的传播和进化关系上显得尤为重要。
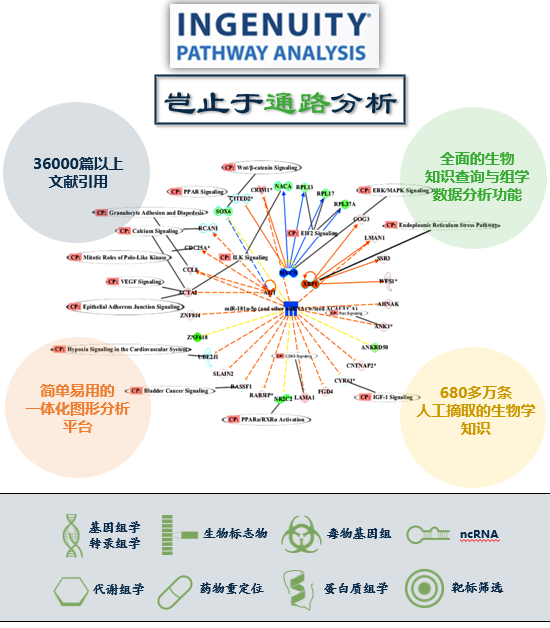
▲ 著名的生物通路分析软件方案Ingenuity Pathway Analysis(IPA)通过其自有的Ingenuity Knowledge Base(IKB)知识库,为基因组、转录组、蛋白质组和代谢组的提供更深刻的生物学洞见。
IPA可以在研究数据中通过建模,分析并理解复杂生物和化学系统。预测下游的效应,上游调控,发现分子间的因果关系。寻找新的潜在生物标志物,药物靶点。探索COVID-19疾病的生物学机制和帮助新药研发,药物机理研究。
通过IPA,对自己实验获得的数据洞察其中的生物学意义,也可将自己的数据和已经公开发表的数据集进行匹配,比较验证。
背景介绍
CLC Genomics Workbench
CLC Genomics Workbench 是一款生物信息学综合分析软件,可用于基因组学,转录组学和表观遗传组学的数据分析。支持MAC OS X,Windows和Linux平台。软件通过ISO 9001:2015认证,可分析和显示所有主流测序仪产生的测序数据。友好而直观的操作界面,只需要鼠标点击就可以完成二代测序的相关分析,方便科研工作者将更多的时间投入研究领域。目前在新冠研究领域已在多个顶级期刊应用CLC发表论文,比如NEJM[1], The Lancet[2]等。
应用1. 重测序分析——可用于新冠病毒序列研究
CLC Genomics Workbench支持用于高精度检测和比较遗传变异的完整重测序流程。包括最开始的QC,Read mapping和最后的Local realignment用来显著提高精度和降低假阳性。检测的变异主要涉及SNV,MNV 和InDels,并进行相关注释,过滤和功能分析。除此之外,软件还支持Genome Browser,可以将各个分析步骤的结果在同一界面进行可视化操作。
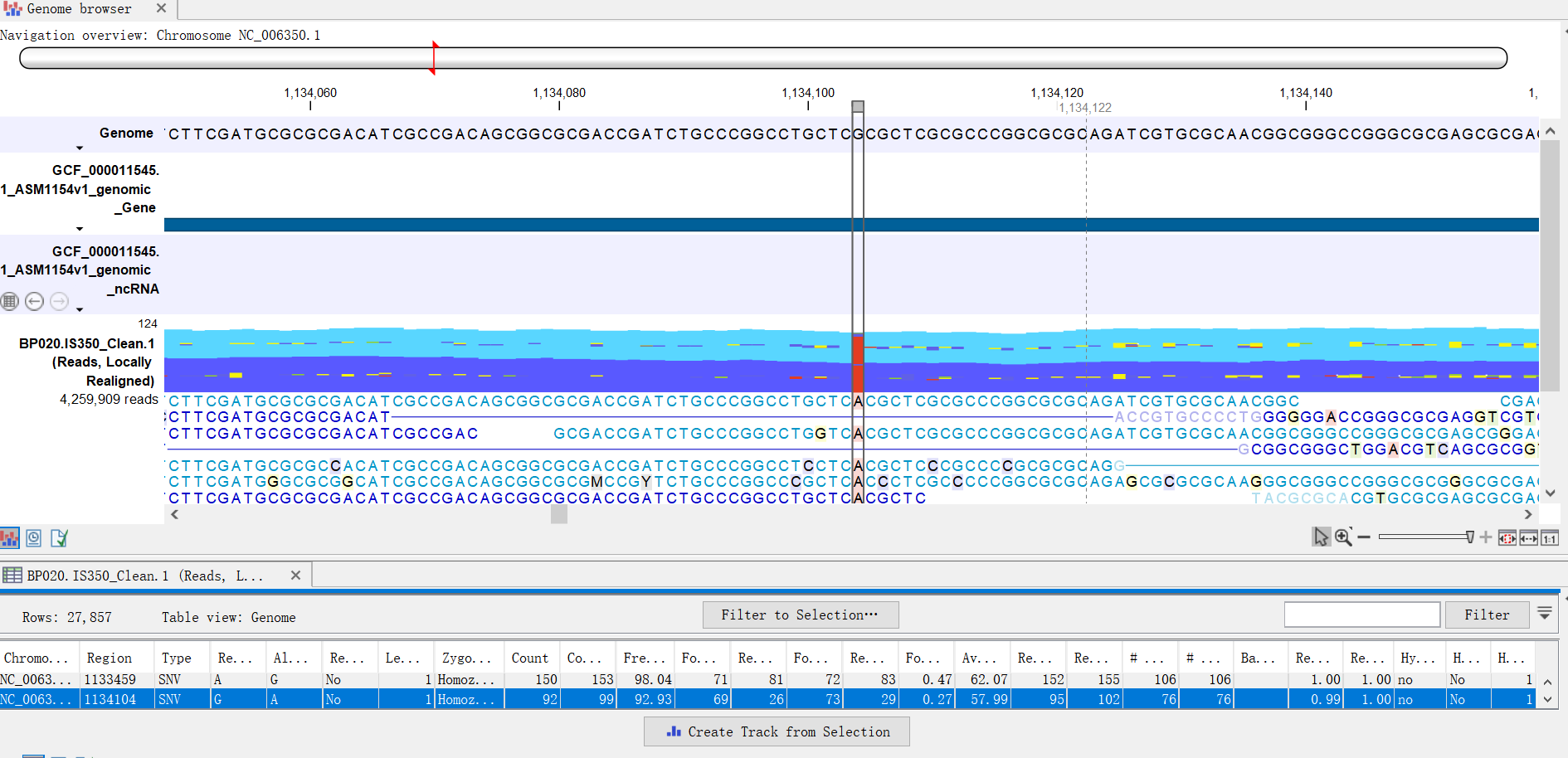
图1 使用Genome Browser 来查看感兴趣的SNV在Reads和注释中的详细情况
应用2. 宏基因组——新冠病毒遗传进化分析,功能注释,组装
CLC具有一套完整的微生物测序分析。 以前收费的插件CLC Microbial Genomics Module和CLC Genome Finishing Module现在完全限时免费啦! 主要涉及微生物的种群鉴定MLST,抗生素耐药性分析,宏基因组的拼接,注释和比较分析,微生物种群分析(16S,18S和ITS扩增子分析,αβ多样性分析等)
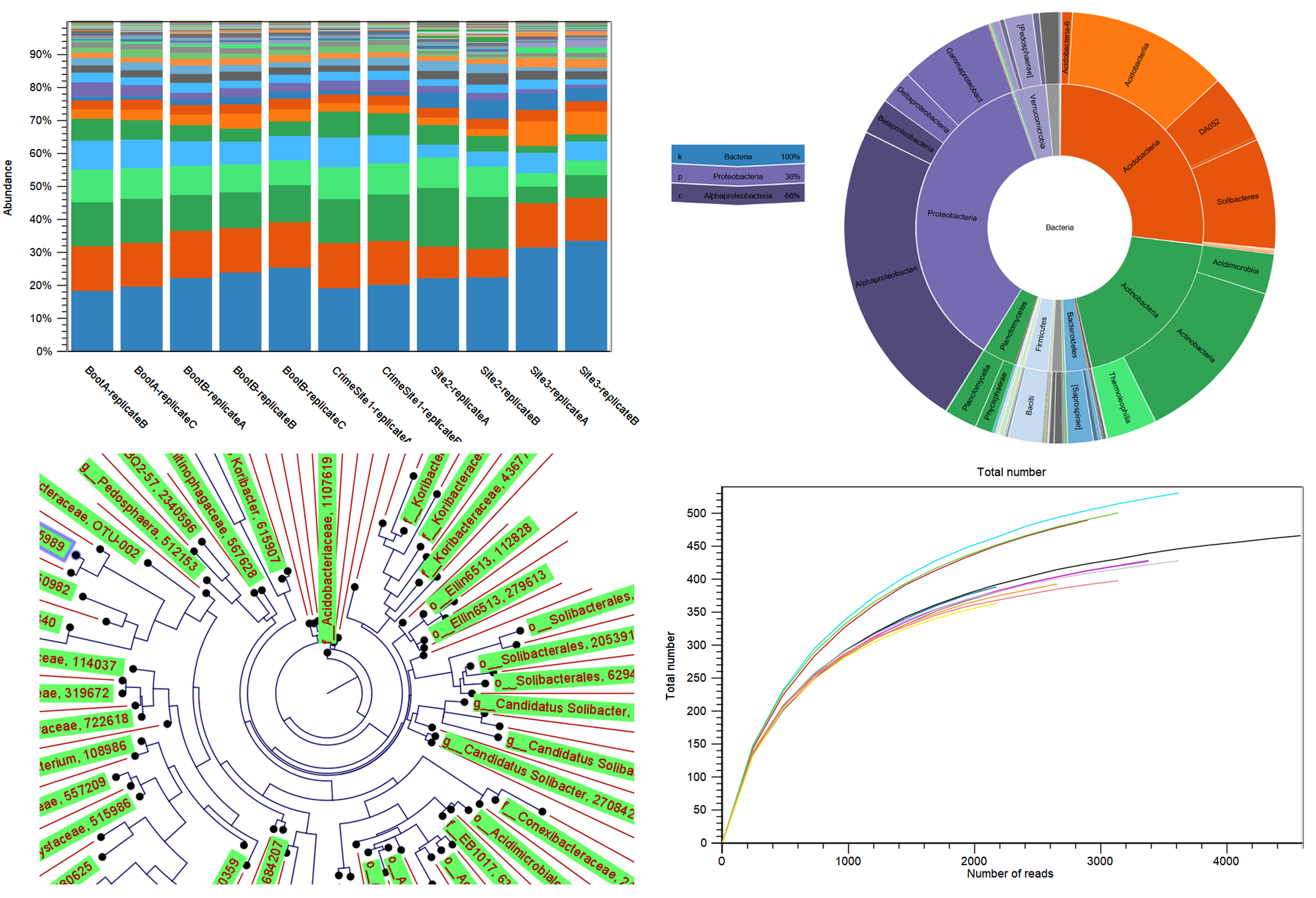
图2 宏基因组相关分析,从左到右为富集分析,太阳图,遗传树和α多样性分析
应用3. RNA-seq——新冠病人转录组数据分析
RNA-seq的分析常用来检测差异基因,CLC在进行数据的质控后,可计算基因和转录本的表达值,比如Total Counts, Unique Counts, TPM和RPKM,然后将统计结果进行可视化选项进行可视化:火山图,热图,PCA图和维恩图等。
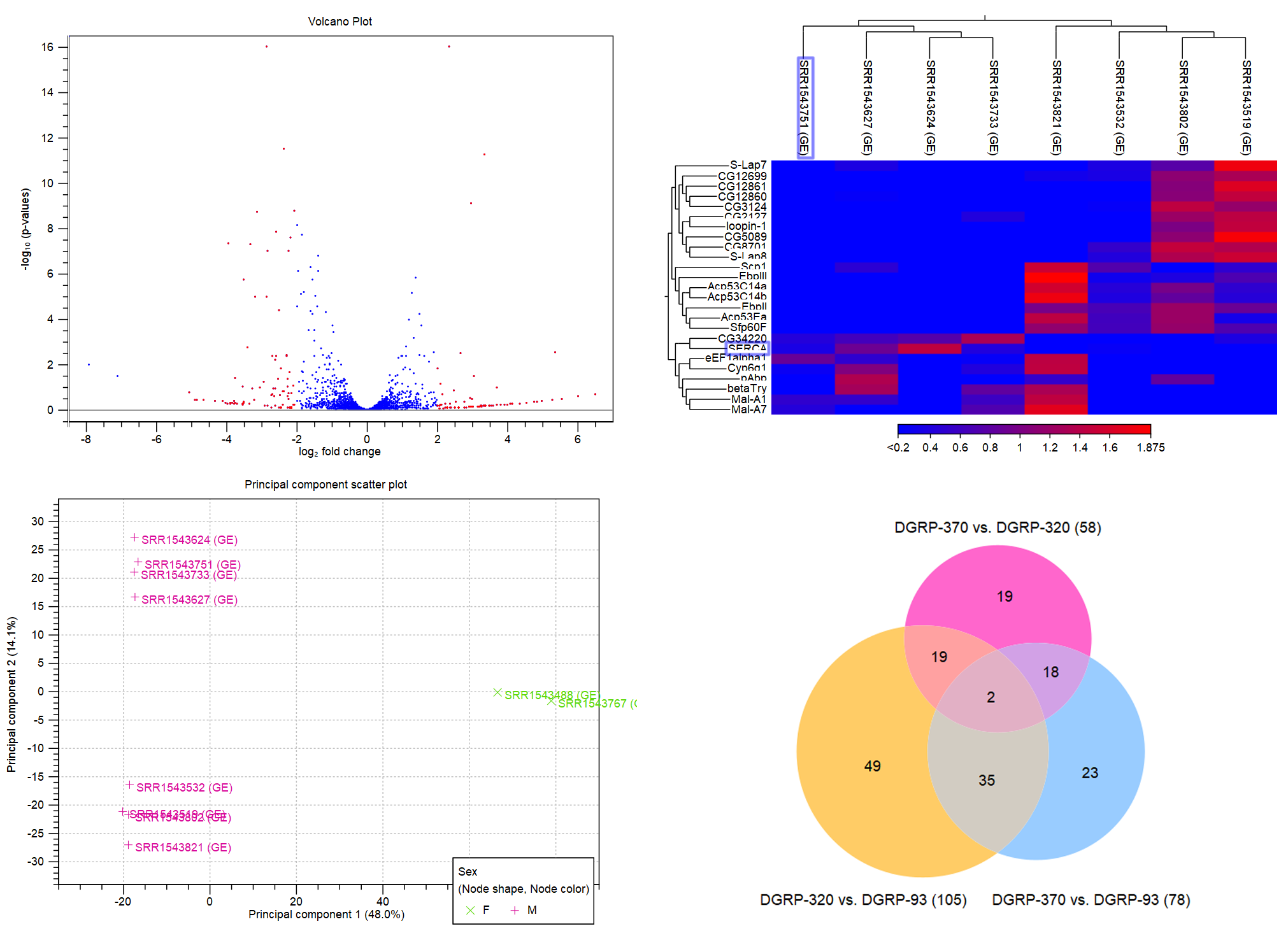
图3 从左到右依次为RNA-seq分析的火山图,热图,PCA图和维恩图
Ingenuity Pathway Analysis(IPA)
IPA作为生物通路分析行业的领导者之一,搭建了Ingenuity Knowledge Base(IKB)知识库。该数据库自1998年起开始构建,着重收集生命科学、医学各领域权威杂志全中文中的生物信息,目前含有近700万条生物学知识,并与多个权威数据库合作,综合其他权威生物学知识,保证IPA软件提供的每条信息都有高影响因子的文献支持,从而提供正确详尽的生物学数据分析解决方案。
目前SARS-CoV-2的研究重点已经从病毒本身的检测和临床治疗手段开发,转移到致病机制的揭示和潜在治疗靶点的寻找上,IPA两个不同导向的解决方案助力SARS-CoV-2研究。
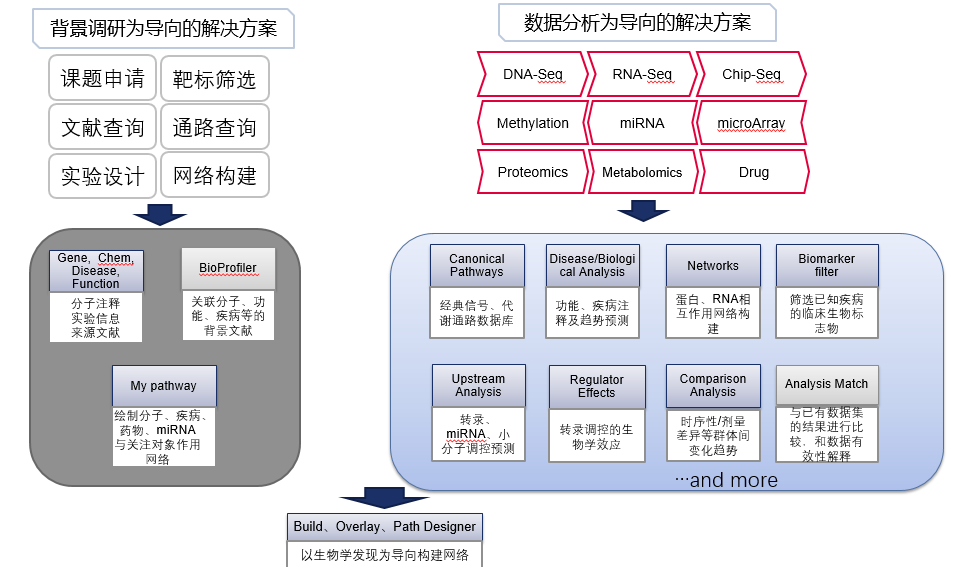
导向1: My Pathway构建——以问题为导向
基于IPA的IKB知识库,使用IPA查询功能,避免耗时费力的人工文献查阅筛选工作,加速新冠研究项目设计效率。研究人员可以快速找到SARS-CoV-2相关疾病的分子信息,并对其进行筛选;也可以寻找感兴趣的分子之间的关系,构建互作网络;还可以对网络进行拓展,整合和美化。且无需我们提供任何实验数据,可以为背景调研,项目开题提供强大的信息支持。
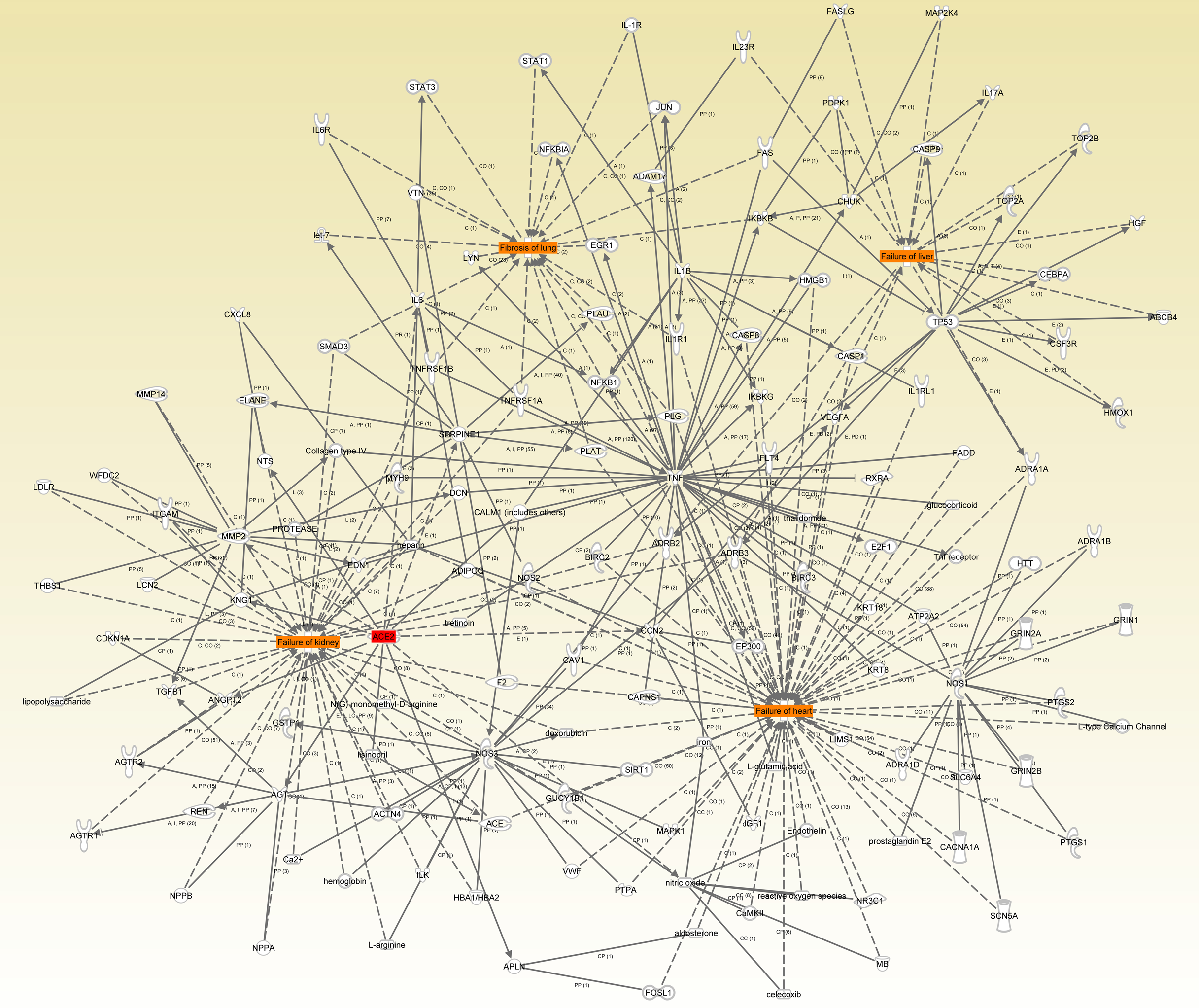
基于IPA构建的新冠相关蛋白互作网络图
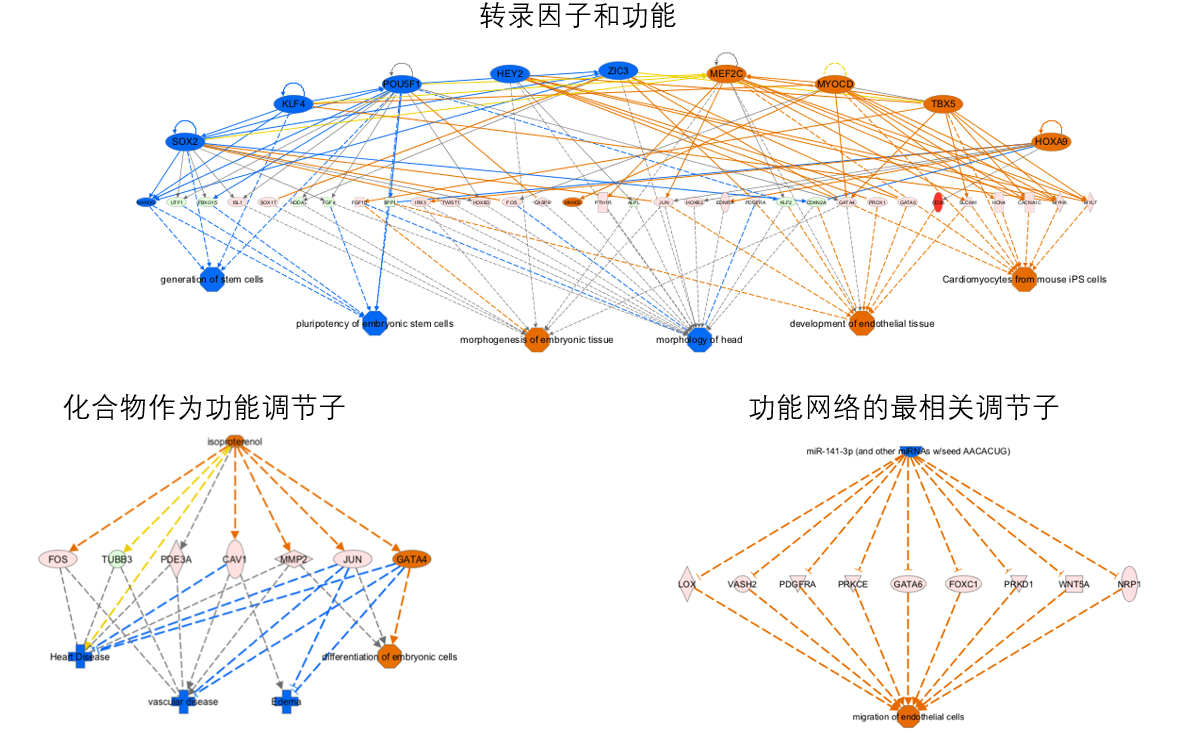
导向2:疾病功能、经典信号通路,互作网络,上游调控因果网络等——数据为导向
IPA可以针对基因组学,蛋白组学或代谢组学实验数据,进行一站式的数据分析和深度挖掘,其中包括疾病功能相关性分析,生物学下游效应分析,经典通路相关性分析,通路活性效应预测,转录调控分析,转录因子互作分析,非经典信号通路预测,互作网络分析等等。所有分析结果均可提供原始文献出处,所有结果均保证来源于高影响因子的文献中的验证实验结果。帮助我们寻找可能的疾病标志物,药物作用靶标,对于SARS-CoV-2的致病机理,和新药研发提供强大助力!
IPA的一站式组学数据分析解读方案,采用每周统一更新的背景知识库。在大大提高分析效率的同时,也避免了传统生物通路分析流程会出现的,在各个数据库平台间切换时,因软件算法、背景数据库质控标准或更新不一致引起的误差。
限时免费CLC Genomics Workbench Pro & Ingenuity Pathway analysis
时间: 从即日起至2020-6-15
活动须知:
1、活动时间:即日起至6月15日
2、申请条件:
a) 科研院校PI发送关于软件的试用申请(务必与新冠研究相关)
b) 关注康昱盛微信公众号并转发“史无前例!助力新冠, IPA & CLC软件限时免费使用!!”本条微信至朋友圈,集齐11个赞,截图。
将试用申请及朋友圈集赞截图,发至marketing@cloudscientific.com。
收到您的申请后,我们将在一个工作日联系您。
本活动最终解释权归于上海康昱盛信息科技有限公司和凯杰企业管理(上海)有限公司所有。
参考文献
[1] A Novel Coronavirus from Patients with Pneumonia in China, 2019. NEJM, DOI: 10.1056/NEJMoa2001017
[2] Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. The Lancet, DOI: https://doi.org/10.1016/S0140-6736(20)30251-8

