
Mascot Distiller 2.7 正式发布
Mascot用户们应该都知道,Mascot产品线的三大软件,它们的功能各有不同。它们的组合可以实现多种蛋白质谱的数据分析,其中主要执行蛋白鉴定的是Mascot Server,读取原始数据、进行谱峰提取,进行基于MS1定量的是Mascot Distiller,Mascot Daemon则作为一个整合Mascot Server和Mascot Distiller的平台,提供批处理递交分析任务的功能。告诉大家一个好消息,Mascot Distiller已经提前更新到了2.7版本。
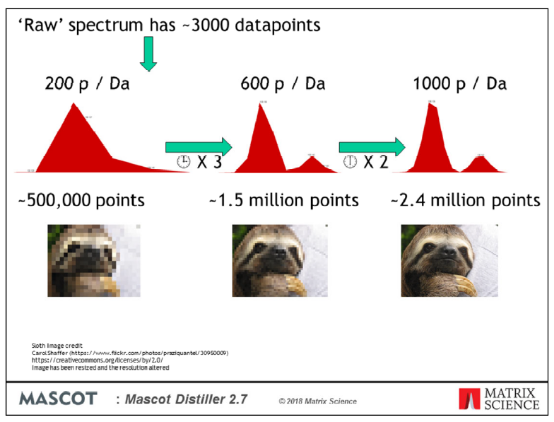
Mascot Distiller2.7版本的主要新特性是改变了在原始廓形数据集中谱峰检测工作方式,这些数据集已被保存为稀疏数据或压缩数据,并伴随着零值删除。咦?表示读不懂?没关系,后面我们慢慢道来。先回到实际问题,Thermo 和Sciex的数据常常保存为profile形式,Mascot Distiller 2.7告别了老版本对一级质谱的re-gridding处理。在之前的版本中,进行谱峰提取要求profile数据是线性质量尺度,数据点在m/z轴上分布均匀。然而,一些profile数据是通过减少0信号强度的数据点来压缩的。因此,在较早版本Mascot Distiller能够处理这些扫描之前,有必要将这些数据转换为分布均匀的m/z值,并且对原始数据点不均匀分布或不确定m/z数值的数据点的扫描进行加和,这一过程这被称为Re-gridding。增加Re-gridding 的数值将增加数据处理时间,所以一般将该数值设置到足够保证最窄峰的峰型。
举个例子,在re-gridding之前,如果你的profile raw data经过了压缩或者“稀疏”(与稀疏矩阵概念有关),那么数据就看起来像左下图这样,非连续并且是不同的数据点组成了峰,并且,峰之间的0值是缺失的。同一个数据集,如果我们设置了每个Dalton的间距为600个点,进行re-gridding,那么数据就看起来像右下图这样,数据点均匀地分布在m/z轴上,并且零值也有数据点,这样我们就有了连续数据。这就是老版本Mascot Distiller 在处理一级谱时做的工作。
然而,在高分辨质谱数据,我们如果为了得到更“保真”的谱峰而提高re-gridding点数的设置,通过Distiller2.6的处理会得到更多的数据点,这也就是为什么过去Distiller处理数据那么慢的原因了。
在Mascot Distiller2.7中,谱峰检测直接根据原始的profile数据工作,而不需要re-gridding。简单地说,就是数据处理速度更快啦!从根本上,Matrix Science的开发者改进了Distiller的算法,去除了对稀疏数据这种类型进行re-gridding的要求。这样,Distiller也不会遇到之前如果没有设置报告离子峰区段而产生的报告离子峰提取错误的问题了。
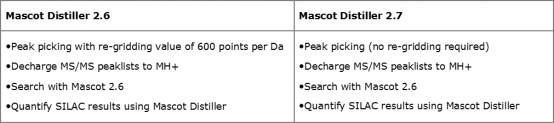
我们这里用一组PRIDE 数据库(PXD004607)已发表的SILAC数据对Mascot Distiller2.6和2.7版本进行了比较测试,计算机的CPU配备的是有点old fashion的Intel Xeon X5650。该数据集有10个原始数据文件,一共120万张谱图。Distiller2.6和2.7的处理方法分别列在下面:
在定量分析阶段,Distiller必须对XIC区域的一级MS扫描进行额外的谱峰检测,按照我们前面所说的Mascot Distiller2.7的谱峰提取算法推想,它的处理速度会有所提高。比较结果如下:

如果数据保存为真实的profile data(常见于Bruker的数据),或者保存为centroid数据格式,那么这种改进则不太明显。
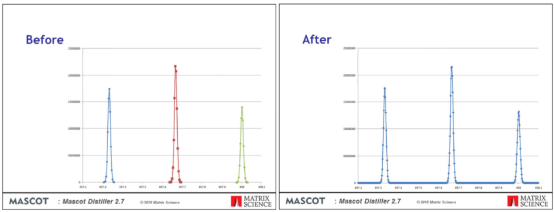
除了提高速度以外,这组数据在同样1%FDR的卡值条件下,Mascot Distiller 2.7在显著性匹配肽段数目上提高了近20%。
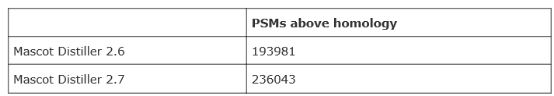
这个结果的产生有三个原因。在Distiller 2.7中,提高了13C谱峰检测和碎片离子电荷状态测定的准确性。我们还看到了谱峰列表中,许多离子的分数比Distiller 2.6得到的更好。这是因为Distiller 2.7生成了包含较少噪音峰的“更干净”的谱峰列表,提供了更好的信噪比。在某些时候,这会使匹配的分数高于显著性阈值。
如果您对于Mascot Distiller 2.7版本的更新感兴趣,欢迎联系康昱盛,可获取Matrix Science 在2018 ASMS大会上对新版本软件详细介绍的PPT文件。


